Learning molecular dynamics: GICnet
分子总是在运动,这种运动可以用 MD 模拟,但需要很多步骤来评估,而且速度很慢。我们可以用 机器学习势 来加速这个过程,更快地计算力。然而,机器学习势仍然不能解决动力学的基本问题:它是迭代的,步骤不能并行化,计算质量取决于时间步长,轨迹是离散的而不是光滑的。
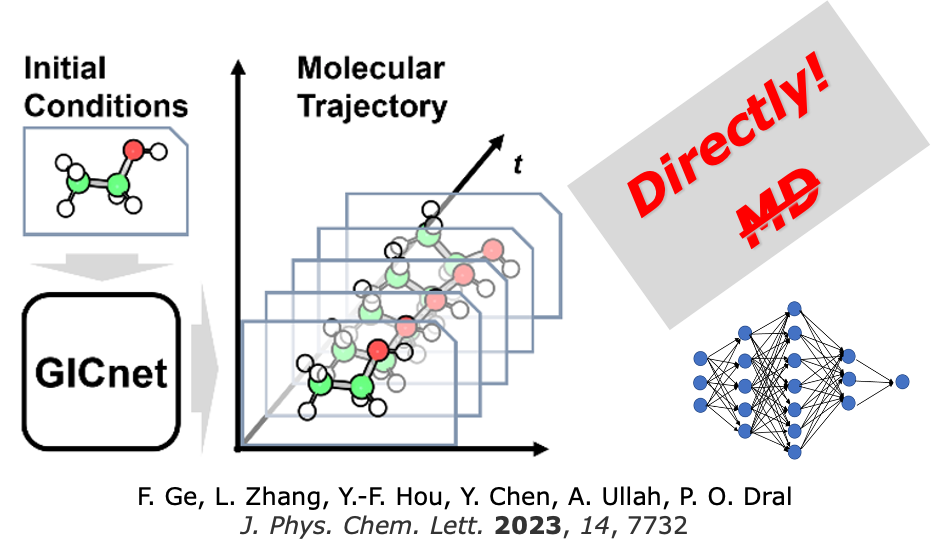
正因如此,我们于 2022年3月 提出了一种新概念,即通过 4D时空原子人工智能模型 (简称4D模型)直接学习动力学的新概念。这一构想看似简单,实现起来并不容易:我们预测核坐标是时间的连续函数!由于不同时间步长的预测无需按顺序计算,因此我们可以并行处理这些预测。轨迹也可以获得任意高分辨率。
GICnet
我们成功地完成了一项艰巨的任务,即创建一个深度学习模型来学习动态并快速预测轨迹。这个模型被称为GICnet,并于2023年在 JPCL上发表 。实际上,GICnet模型是四维时空中分子和化学反应的分析表示!
令人惊讶的是,我们的模型可以学习不同复杂分子的动力学,甚至可以学习偶氮苯的顺反异构化动力学。使用GICnet可以比使用机器学习势更快地获得轨迹。
详情请参阅我们的论文(如果您使用GICnet,请同时引用):
Fuchun Ge, Lina Zhang, Yi-Fan Hou, Yuxinxin Chen, Arif Ullah, Pavlo O. Dral*. Four-dimensional-spacetime atomistic artificial intelligence models. J. Phys. Chem. Lett. 2023, 14, 7732–7743. DOI: 10.1021/acs.jpclett.3c01592.
备注
在本教程中,我们只讨论Python API。对于输入文件/命令行使用,只有特定版本的MLatom支持GICnet,可以在 GitHub 中找到。
现在,让我们进入在MLatom中使用GICnet的说明部分。
预备条件
MLatom 3.8.0或者更新版本tensorflow v2.17.0(对其他版本不作保证)
训练GICnet模型
这里我们选用乙醇分子。
首先,我们需要生成一些轨迹作为训练数据。请查阅本 教程 在MLatom中运行MD。
from mlatom.GICnet import GICnet
mol = ml.molecule.from_smiles_string('CCO')
molDB = ml.generate_initial_conditions(
molecule = mol,
generation_method = 'random',
initial_temperature = 300,
number_of_initial_conditions = 8
)
ani = ml.models.methods('ANI-1ccx',tree_node=False)
trajs = [
ml.md(
model=ani,
molecule=mol,
time_step=0.05,
ensemble='NVE',
maximum_propagation_time=10000,
).molecular_trajectory
]
然后,我们可以使用这些轨迹来训练GICnet模型:
model = GICnet(
model_file='GICnet', # the name of the model file (it's a folder)
hyperparameters={
'ICidx': 'ethanol.ic', # a file that contains the definition of the internal coordinates for ethanol
'tc': 10.0, # the time cutoff
'batchSize3D': 1024, # the batch size for the built-in MLP model
'batchSize4D': 1024, # the batch size for the 4D model
'maxEpoch3D': 1024, # the maximum number of epochs of MLP model training
'maxEpoch4D': 1024, # the maximum number of epochs of 4D model training
}
)
model.train(trajs)
由于GICnet使用内部坐标来描述分子,因此我们需要提供坐标系统的定义。更多的细节可以在我们的论文中找到,这里是乙醇的一个例子,其定义保存在文件 ethanol.ic 中:
1 0 -1 -2
2 1 0 -1
3 1 2 0
4 1 2 3
5 1 2 3
6 2 1 3
7 2 1 3
8 2 1 3
9 3 1 2
训练数据将实时打印在stdout中;
... ...
----------------------------------------------------
epoch 5 1/1 t_epoch: 0.1
validate: 0.000650
best: 0.000650
----------------------------------------------------
epoch 6 1/1 t_epoch: 0.1
validate: 0.000599
best: 0.000599
----------------------------------------------------
... ...
----------------------------------------------------
epoch 1 1/1 t_epoch: 1.9
D A DA Ep/Ek
train: 0.045998 0.049149 0.036743 0.005203
d/dt 0.009045 0.009566 0.008752 0.000000
Loss 0.842420
validate: 0.031084 0.024374 0.019558 0.001658
d/dt 0.013125 0.010855 0.009795 0.000000
Loss 0.821911
best: 0.031084 0.024374 0.019558 0.001658
d/dt 0.013125 0.010855 0.009795 0.000000
Loss 0.821911
----------------------------------------------------
epoch 2 1/1 t_epoch: 2.0
D A DA Ep/Ek
train: 0.036901 0.049857 0.033790 0.005391
d/dt 0.008148 0.008873 0.008209 0.000000
Loss 0.781977
validate: 0.034426 0.029851 0.021169 0.003375
d/dt 0.012806 0.009719 0.007115 0.000000
Loss 0.752215
best: 0.034426 0.029851 0.021169 0.003375
d/dt 0.012806 0.009719 0.007115 0.000000
Loss 0.752215
----------------------------------------------------
... ...
用GICnet模型传播轨迹
一旦你有了一个GICnet模型,你就可以用它以闪电般的速度传播动力学轨迹。
from mlatom.GICnet import GICnet
mol=ml.molecule.from_smiles_string('CCO')
mol= ml.generate_initial_conditions(molecule = mol,
generation_method = 'random',
initial_temperature=300)[0]
model = GICnet(model_file='GICnet')
# options are similar to normal MD
dyn = model.propagate(molecule=mol,
maximum_propagation_time=20000, # fs
time_step=0.1 # fs
)
我们极力推荐训练多个模型,并通过它们的集成传播动力学轨迹,以提高预测的稳定性:
model = GICnet(model_file=['GICnet_0',
'GICnet_1',
'GICnet_2',
'GICnet_3'])
dyn = model.propagate(molecule=mol,
maximum_propagation_time=20000,
time_step=0.1)